
基于生物信息学探究动脉粥样硬化与糖尿病肾病共病遗传机制研究
摘要:目的:应用多种生物信息学方法探究动脉粥样硬化与糖尿病肾病共病的遗传机制。方法:本研究首先通过检索GEO数据库获得动脉粥样硬化与糖尿病肾病的训练数据集和验证数据集;然后分别将两种疾病训练集的数据进行差异分析获得两种疾病和对照组的差异基因并取二者交集以得到共病差异基因;接下来将共病差异基因构建蛋白质相互作用网络并使用Cytoscape的插件构建核心网络以筛选共病核心基因;本研究还将共病核心基因进行富集分析并找出其关联的转录因子以构建转录因子调控网络;最后进行验证分析以对结果的准确性和稳健性进行确认。结果:本研究获得动脉粥样硬化和糖尿病肾病的训练和验证数据集各1个;通过差异分析得到动脉粥样硬化组差异基因517个,糖尿病肾病组差异基因1430个,将二者取交集后得出共病基因64个;通过构建蛋白相互作用网络得到MS4A6A、CFH、CD163、POSTN、MS4A4A、FOLR2等15个共病核心基因的子网络;通过富集分析得到共病核心基因主要涉及体液免疫调节和应答、补体激活、吞噬作用等机制并得到SPI1、ETS2、CEBPA、JUN等4个对共病核心基因起调控作用的转录因子以及其调控网络;最后验证了所得共病核心基因和转录因子大部分在验证组存在统计学意义的差异。结论:本研究鉴定出了MS4A6A、CFH、CD163、POSTN、MS4A4A等15个动脉粥样硬化与糖尿病肾病共病的核心基因,这些核心基因主要涉及体液免疫调节和应答、补体激活、吞噬作用等机制。
关键词:基因、遗传、共病遗传、聚类分析、PPI网络、大数据
动脉粥样硬化(Atherosclerosis, AS)是指从内膜开始,局部脂质和复合糖类积聚、纤维组织增生和钙质沉着形成斑块从而导致动脉管壁增厚变硬、失去弹性和管腔缩小为特点的疾病[1],是冠心病、缺血性中风等多种疾病的危险因素[2]。其发病率为2.8%,并且呈上升趋势,给家庭、社会造成了巨大的经济负担[3]。糖尿病肾病(Diabetic Nephropathy, DN)是糖尿病(Diabetes Mellitus, DM)的一种微血管并发症,其特点是持续的蛋白尿、肾功能的进行性下降、心血管疾病和死亡率的风险增加,有40%的DM患者可能发展为DN[4-8],而DN是全球终末期肾病的主要原因,因而DN也是重大的公共卫生问题[9]。
研究表明AS与DN存在密切的相关关系,首先DM是AS与DN的独立危险因素,其次二者有共同的病理过程,即内皮血管扩张剂一氧化氮(NO) 的生物利用度降低,伴有炎症和氧化应激升高[10]。此外,临床横断面研究表明DN是严重AS的最重要独立危险因素[11]。但是AS和DN具体的遗传学机制仍有待进一步研究。鉴于利用微阵列和生物信息学技术的全基因组转录组分析可以识别疾病进展的关键靶点,并为疾病的发病机制和分子分类提供见解[12],因此我们应用生物信息学方法分析AS与DN共病遗传机制, 从而为临床及进一步研究提供参考。
1.资料与方法
1.1 疾病数据集获取与处理
以“Atherosclerosis”“Diabetic nephropathy”为关键词,并限定物种为智人,数据类型为基因表达数据,在GEO(Gene Expression Omnibus, 444https://www.ncbi.nlm.nih.gov/geo/)数据库中检索以分别获得动脉粥样硬化和糖尿病肾病的包含训练集和验证集基因表达数据。
1.2 疾病差异基因与共病交集基因
将动脉粥样硬化及糖尿病肾病的训练集数据分别分为对照组及疾病组,然后使用R语言的“limma”包进行差异分析,按照|log2(FC)|>1及校正后P<0.05进行筛选,从而获得动脉粥样硬化患者与对照患者的差异基因以及糖尿病肾病与对照患者的差异基因,并表示为热图和火山图。然后将两者使用Venn图取交集,获得动脉粥样硬化与糖尿病肾病共病的差异基因。
1.3 富集分析与蛋白质相互作用网络构建
使用动脉粥样硬化与糖尿病肾病的共病基因使用R语言进行GO及KEGG富集分析以分析其生物功能及涉及通路;然后将共病基因导入Cytoscape的String插件构建PPI网络,并使用MCODE、Cytohubba等分析PPI网络从而筛选出核心共病基因;接下来,将核心共病基因导入GeneMANIA平台(https:// genemania.org/)以找出与核心基因具有蛋白互作关系的基因,从而扩展我们的结果;最后使用核心基因再次进行GO和KEGG富集分析以聚焦结果。
1.4 疾病验证数据集验证核心基因
我们将动脉粥样硬化和糖尿病肾病的验证集分别分为对照组和疾病组,然后依次将每一个核心基因在对照组和疾病组中的表达进行差异分析以验证我们筛选的核心基因结果,其中P<0.05为存在统计学差异,结果以小提琴图表示。
1.5 转录因子富集分析、调控网络构建与验证
使用TRUST 平台(https://www.grnpedia. org/trrust/)对核心基因进行转录因子富集分析;然后使用Cytoscape软件以转录因子、核心基因为节点,其相互关系为边构建转录因子和核心基因的网络,即转录因子对核心基因调控作用的网络。
1.6 统计分析
本研究使用R语言及Cytoscape进行统计学分析。对于差异分析,我们设定|log2(FC)|>1及校正后P< 0.05为具有统计学差异;对于PPI网络筛选,我们设置Degree cutoff=2, Node score Cutoff=0.2,K-core=2,Max. Depth=100。对于富集分析我们都以校正后的P<0.05为具有统计学差异。
2.结果
2.1 动脉粥样硬化与糖尿病肾病数据集获取
通过检索GEO数据库后,本研究获得动脉粥样硬化数据集2个,其中训练集 (GSE28829) 包含13个对照组样本,16个动脉粥样硬化样本;验证集 (GSE100927) 包含36个对照组样本,68个动脉粥样硬化样本;本研究获得糖尿病肾病数据集2个,其中训练集 (GSE96804) 包含20个对照组样本,41个糖尿病肾病样本;验证集 (GSE30122) 包含40个对照组样本,19个糖尿病肾病样本。
2.2 疾病差异基因与共病交集基因结果
通过对训练组进行差异分析,我们得到动脉粥样硬化组有517个差异基因,其中KCNK17、CASQ2、MYBL2、SBSPON和INPP5A等在对照组中上调,IGJ、IGLC1、IGLL3P、HP和CCL19等在动脉粥样硬化组上调,如图1A、1B;糖尿病肾病组有1430个差异基因,其中DEFA1B、DEFA3、EGFR、FOS和DUSP1等在对照组上调,C1QC、FPR3、MARCKS、FN1和C3等在糖尿病肾病组上调,如图1C、1D。通过将动脉粥样硬化差异基因与糖尿病肾病差异基因取交集,我们得到50个共病交集上调基因与14个共病交集下调基因,如图1E、1F。
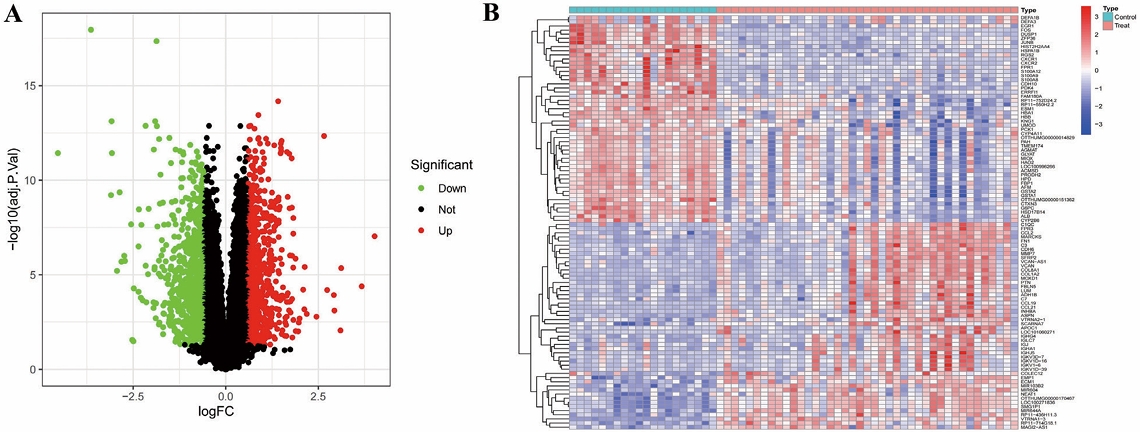
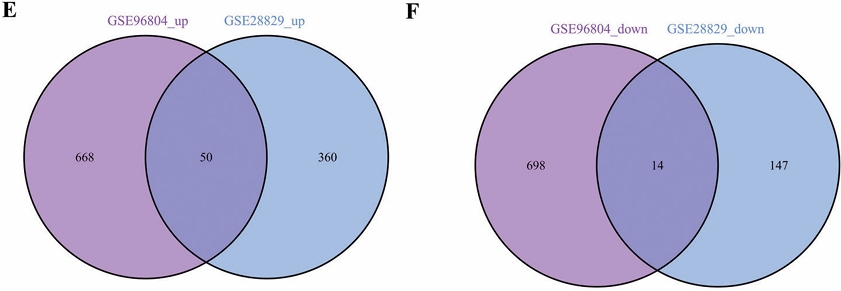
图1 疾病差异基因与共病交集基因结果 (A) 动脉粥样硬化训练集差异分析火山图;(B) 动脉粥样硬化训练集差异分析热图;(C) 糖尿病肾病训练集差异分析火山图;(D) 糖尿病肾病训练集差异分析热图;(E) 动脉粥样硬化与糖尿病肾病上调差异基因韦恩图;(F) 动脉粥样硬化与糖尿病肾病下调差异基因韦恩图。
2.3 富集分析与PPI网络构建及分析结果
我们将共病交集差异基因进行富集分析,得到补体激活替代通路、免疫反应激活、对细胞粘附的负调控作用、树突状细胞凋亡过程、树突状细胞凋亡过程的调控、白细胞−细胞粘附的负调控、细胞对肿瘤坏死因子的反应和体液免疫反应等免疫炎症相关的GO生物过程项;含有胶原蛋白的细胞外基质、胶原三聚体、血微粒、空泡腔和血浆脂蛋白颗粒等GO细胞成分项;货物受体活性、细胞外基质结构成分、调理素结合、补体结合和肽酶调节活性等GO分子功能项;补充和凝血级联、吞噬小体、NF−kappaB信号通路和Wnt信号通路等KEGG通路项,具体见图2A、2B。
通过构建PPI网络(图2C),我们得到LGALS9、CTSC、CCL19、NCKAP1L和C1QC等64个差异基因的蛋白互作关系,其中除RBP4之外,其他差异基因均为正调控;通过MCODE进行聚类分析,我们得到2个聚类(图2D),其中C1包括VSIG4、MSR1、MRC1、C1QC、 FPR3、FOLR2、MS4A4A、MS4A6A;C2包括CCL21、CCL19、C7;通过Cytohubba进行核心PPI网络进行筛选(图2E),我们得到15个核心基因的子网络,涉及MS4A6A、CFH、CD163、POSTN、MS4A4A、FOLR2、LUM、CD93、C1QC、VCAM1、CTSK、MSR1、 VSIG4、MRC1、C3等。
通过使用子网络的核心基因构建GeneMANIA网络(图2F),我们得到TRAPPC2L、SIGLEC1、 C1QB、CFB和C1QB等20个与核心基因存在共表达、生理相互作用、共定位、共享蛋白结构域、遗传相互作用等关系的关联基因,从网络的密度来看,它们之间有较密切的相关性。这些基因之间的协同功能主要表现为:体液免疫调节和应答、补体激活、吞噬作用等。通过将核心基因再次进行富集分析(如图2G、2H),我们得到的结果基本与差异基因的富集分析结果相同,表明了我们得到的体液免疫、补体激活等机制的结果具有稳健性。
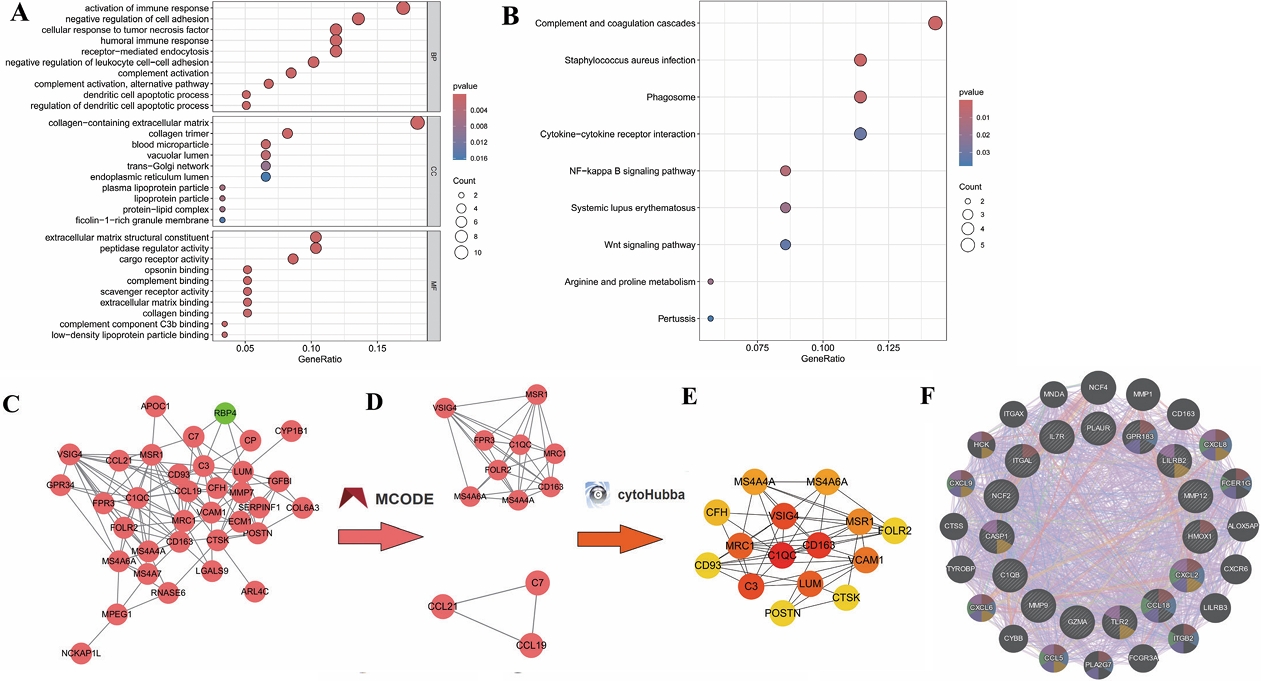

图2 差异基因富集分析与PPI网络构建及分析结果 (A) 共病差异基因GO富集分析气泡图;(B)共病差异基因KEGG富集分析气泡图;(C) 共病差异基因PPI网络;(D) PPI网络聚类分析;(E)PPI网络核心子网络分析;(F)核心基因的GeneMANIA网络;(G)核心基因GO富集分析圈图;(H)核心基因KEGG富集分析圈图
2.4 疾病验证数据集验证核心基因结果
通过使用GEO验证集将核心基因在疾病组与控制组的表达差异进行差异分析,结果可知对于动脉粥样硬化验证集,15个核心基因中除CFH以外,有14个存在统计学差异,具体见 图3A;对于糖尿病肾病验证集。15个核心基因中除 CD93、CFH和POSTN等3个基因无统计学意义外,其余12个核心基因均有统计学差异,具体见图3B。
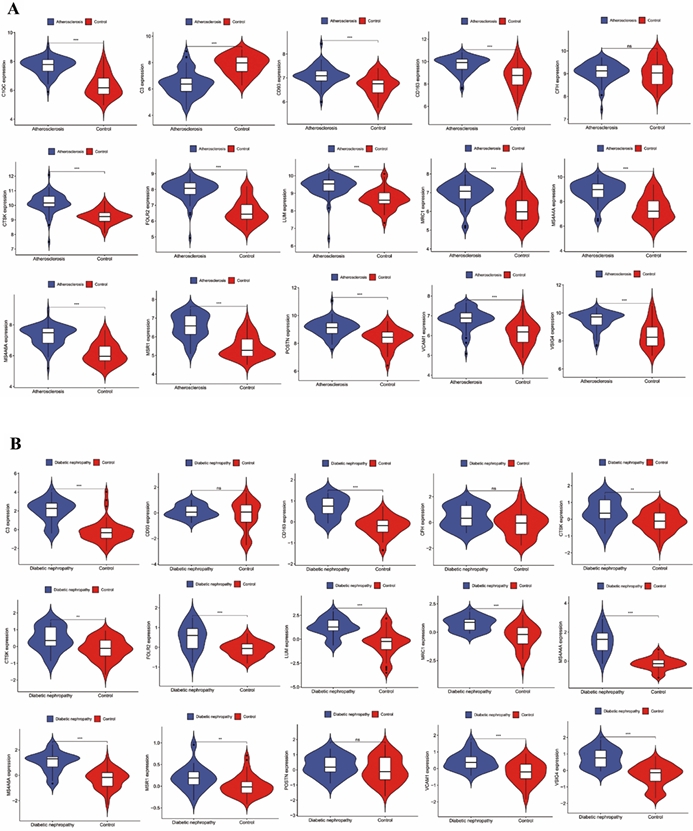
图3 验证集验证核心基因表达差异分析结果(A)动脉粥样硬化验证集核心基因表达差异分析小提琴图;(B)糖尿病肾病验证集核心基因表达差异分析小提琴图
2.5 转录因子富集分析、调控网络构建与验证结果
我们通过进行转录因子富集分析从而得到与核心基因存在调控关系的转录因子有SPI1、 ETS2、CEBPA、JUN等4个,它们之间的调控关系如图4A所示。通过对这些转录因子进行在疾病组和控制组之间的表达差异分析,我们可知在动脉粥样硬化组有CEBPA和SPI1表达存在统计学差异(图4B),在糖尿病肾病组有JUN的表达存在统计学差异(图4C)。
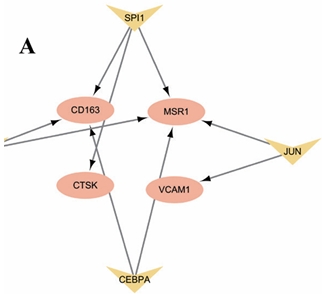
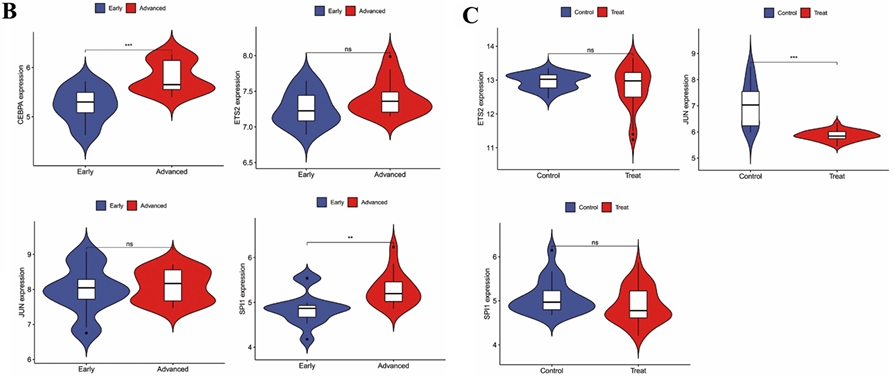
图4 转录因子调控网络构建与验证结果(A)转录因子调控核心基因网络;(B)调控转录因子动脉粥样硬化数据集差异表达分析小提琴图;(C)调控转录因子糖尿病肾病数据集差异表达分析小提琴图
3.结论
AS与DN均为常见循环系统疾病,二者在临床常同时出现,且均与内皮功能障碍、脂质代谢、免疫炎症、氧化应激和胰岛素抵抗等机制有关,因而具有广泛的相关性[13-18]。
我们通过构建PPI网络得到的核心基因中:MS4A6A被研究表明与DN的进展有关,并且与AS斑块形成有关[19, 20];CFH被动物实验证实其可以通过补体C34相关性[21, 22];CD163是巨噬细胞转化的关键受体,而该受体是DN的潜 在治疗靶点;而对于AS,有观点认为CD163具有AS保护作用,但更进一步的研究则显示该受体可促进AS[23, 24];POSTN编码估骨膜蛋白,该蛋白已被实验证明可通过激活外膜成纤维细胞促进AS外膜重塑并通过累积作用增强DN的肾纤维化[25, 26];MS4A4A在一项人类样本分析中被证明与DN存在负相关,并且是AS发病的枢纽基因[27, 28];FOLR2为选择性激活巨噬细胞相关基因,该基因被证实在AS中上调,但其与DN的关系还有待进一步研究[29];LUM人类肾活检样本中表现为DN明显下调,而在AS中则在突出定位于增厚的内膜基质中,从而表现促进胶原纤维合成的作用[30, 31];CD93在小鼠实验中表明其表达与AS呈负相关,并且是DN的候选生物标志物[32, 33];C1QC在一项基因表达谱分析中经鉴定为AS免疫机制的特征基因,在DN中则被证明可显著增加从而激活补体从而加速DN[34, 35];VCAM1被研究证实其介导的炎症反应是DN发生的主要机制,而其在AS斑块中表达降低[36, 37];CTSK编码一种细胞外基质蛋白降解的介质,可以直接导致AS,而该基因与DN的关系则缺乏进一步证实[38];MSR1编码巨噬细胞清除受体1,该受体影响包括AS在内的多种 症相关疾病和癌症;同时肾小球巨噬细胞可能通过该受体被糖氧化和脂质过氧化产物激活从而在DN中发挥作用[39, 40];VSIG4在动物实验中显示在DN中上调,并被认为是DN进展的重要介 质,同时该基因可以作为AS的血液生物标志物[41, 42];MRC1是M2相关巨噬细胞标志物,其在AS中主要起促进作用,在DN中的作用仍未知[43];C3是补体成分,被认为是DN的促进因素,并且参与了AS的过程[44, 45]。
总体来看这些AS与DN的共病核心基因主要涉及补体、免疫炎症等方面,该结果与GO和KEGG富集分析结果相吻合。而我们分析得到的对共病核心基因起调控作用的转录因子包括SPI1、JUN、ETS2和CEBPA四个,其中SPI1编码一种先锋转录因子,该转录因子对于B细胞的转化尤为重要,并且其诱导髓系祖细胞巨噬细胞分化受到抑制[46];多项研究表明该转录因子涉及DN与AS的发生机制[47, 48]。JUN编码 一种与病毒蛋白高度相似的蛋白质,并直接与特定的靶DNA序列相互作用以调节基因表达[49];该蛋白可加速早期AS发展并与DN的肾损伤呈正相关[50, 51]。ETS2相关研究较少,未发现其与AS及DN的关系。CEBPA是参与脂肪生成与能量稳态的转录因子,其上调会促进AS斑块破裂与DN的发展[52-54]。
综上所述,通过对人类转录组样本进行生物信息学分析,我们鉴定出了MS4A6A、CFH、CD163、POSTN、MS4A4A、FOLR2、LUM、 CD93、C1QC、VCAM1、CTSK、MSR1、VSIG4、MRC1、C3等15个AS与DN共病的核心基因,这些核心基因主要涉及体液免疫调节和应答、补体激活、吞噬作用等机制。同时我们还鉴定出了SPI1、JUN、ETS2和CEBPA等4个共病调控转录因子。该结论可为进一步研究和临床应用提供参考,但这些数据主要是通过公共数据库所得,因而仍需使用外部样本进行分析。
参考文献
[1] 葛均波,徐永健 . 内科学 [M]. 北京 :人民卫生出版社 , 2013:220.
[2] Chow YL, Teh LK, Chyi LH, et al. Lipid Metabolism Genes in Stroke Pathogenesis: The Atherosclerosis[J]. Curr Pharm Des. 2020,26(34):4261-4271.
[3] Robinson JG, Fox KM, Bullano MF, et al. Atherosclerosis profile and incidence of cardiovascular events: a population-based survey[J]. BMC Cardiovasc Disord, 2009,9:46.
[4] Chen W, Li Z, Zhao Y, et al. Global and national burden of atherosclerosis from 1990 to 2019: trend analysis based on the Global Burden of Disease Study 2019[J]. Chin Med J (Engl), 2023,136(20):2442-2450.
[5] Lovic D., Piperidou A., Zografou I., et al. The Growing Epidemic of Diabetes Mellitus[J]. Curr Vasc Pharmacol. 2020, 18(2):104-109.
[6] Umanath K.,Lewis J.B.Update on Diabetic Nephropathy: Core Curriculum 2018[J]. Am J Kidney Dis, 71(6):884-895.
[7] Selby N.M.,Taal M.W. An updated overview of diabetic nephropathy: Diagnosis, prognosis, treatment goals and latest guidelines[J]. Diabetes Obes Metab, 22 Suppl 1:3-15.
[8] Alicic R.Z., Rooney M.T., Tuttle K.R. Diabetic Kidney Disease: Challenges, Progress, and Possibilities. Clin J Am Soc Nephrol. 2017, 12(12):2032-2045.
[9] Qi C., Mao X., Zhang Z., et al. Classification and Differential Diagnosis of Diabetic Nephropathy[J]. J Diabetes Res, 2017:8637138.
[10] Sharma A, Choi J, Sim L, et al. Ameliorating diabetesassociated atherosclerosis and diabetic nephropathy through modulation of soluble guanylate cyclase[J]. Front Cardiovasc Med. 2023,10:1220095.
[11] Barrios C, Pascual J, Otero S, et al. Diabetic nephropathy is an independent factor associated to severe subclinical atheromatous disease[J]. Atherosclerosis, 2015,242(1):37-44.
[12] Xu B., Wang L., Zhan H., et al. Investigation of the Mechanism of Complement System in Diabetic Nephropathy via Bioinformatics Analysis[J]. J Diabetes Res,2021:5546199.
[13] Russo G, Piscitelli P, Giandalia A, et al. Atherogenic dyslipidemia and diabetic nephropathy[J]. J Nephrol, 2020,33(5):1001-1008.
[14] Landis RC, Quimby KR, Greenidge AR. M1/M2 Macrophages in Diabetic Nephropathy: Nrf2/HO-1 as Therapeutic Targets[J]. Curr Pharm Des. 2018,24(20):2241-2249.
[15] Lambie M, Bonomini M, Davies SJ, et al. Insulin resistance in cardiovascular disease, uremia, and peritoneal dialysis[J]. Trends Endocrinol Metab. 2021,32(9):721-730.
[16] Leung WK, Gao L, Siu PM, et al. Diabetic nephropathy and endothelial dysfunction: Current and future therapies, and emerging of vascular imaging for preclinical renal-kinetic study[J]. Life Sci, 2016,166:121-130.
[17] Giacco F, Brownlee M. Oxidative stress and diabetic complications[J]. Circ Res. 2010,107(9):1058-70.
[18] Nomura M, Kasami R, Ohashi M, et al. Significantly higher incidence of carotid atherosclerosis found in Japanese type 2 diabetic patients with early nephropathy[J]. Diabetes Res Clin Pract, 2004,66 Suppl 1:S161-3.
[19] Zeng M, Liu J, Yang W, et al. Identification of key biomarkers in diabetic nephropathy via bioinformatic analysis[J]. J Cell Biochem, 2019,120(5):8676-8688.
[20] Chen Y, You N, Yang C, et al. Helicobacter pylori infection increases the risk of carotid plaque formation: Clinical samples combined with bioinformatics analysis[J]. Heliyon. 2023,9(9):e20037.
[21] Kiss MG, Papac-Miličević N, Porsch F, et al. Cell-autonomous regulation of complement C3 by factor H limits macrophage e f f e r o c y t o s i s a n d e x a c e r b a t e s a t h e r o s c l e r o s i s [ J ] . Immunity,2023,56(8):1809-1824.e10.
[22] Bonomo JA, Palmer ND, Hicks PJ, et al. Complement factor H gene associations with end-stage kidney disease in African Americans[J].Nephrol Dial Transplant. 2014,29(7):1409-14.
[23] Landis RC, Quimby KR, Greenidge AR. M1/M2 Macrophages in Diabetic Nephropathy: Nrf2/HO-1 as Therapeutic Targets[J]. Curr Pharm Des. 2018,24(20):2241-2249.
[24] Guo L, Akahori H, Harari E,et al. CD163+ macrophages p r o m o t e a n g i o g e n e s i s a n d v a s c u l a r p e r m e a b i l i t y accompanied by inflammation in atherosclerosis[J]. J Clin Inves,2018,128(3):1106-1124.
[25] Wang Z, Li G, Li M, et al. Periostin contributes to the adventitial remodeling of atherosclerosis by activating adventitial fibroblasts[J]. Atheroscler Plus, 2022,4450:57-64.
[26] Um JE, Park JT, Nam BY, et al. Periostin-binding DNA aptamer treatment attenuates renal fibrosis under diabetic conditions[J]. Sci Rep, 2017,7(1):8490.
[27] Tao Y, Wei X, Yue Y, et al. Extracellular vesicle-derived AEBP1 mRNA as a novel candidate biomarker for diabetic kidney disease[J]. J Transl Med, 2021,19(1):326.
[28] Zhang J, Zhang L. Bioinformatics approach to identify the influences of SARS-COV2 infections on atherosclerosis[J]. Front Cardiovasc Med. 2022,9:907665.
[29] Bosmans LA, van Tiel CM, Aarts SABM, et al. Myeloid CD40 deficiency reduces atherosclerosis by impairing macrophages' transition into a pro-inflammatory state[J]. Cardiovasc Res, 2023,119(5):1146-1160.
[30] Barwinska D, El-Achkar TM, Melo Ferreira R, et al. Molecular characterization of the human kidney interstitium in health and disease[J]. Sci Adv, 2021,7(7):eabd3359.
[31] Onda M, Ishiwata T, Kawahara K, et al. Expression of lumican in thickened intima and smooth muscle cells in human coronary atherosclerosis[J]. Exp Mol Pathol,2002,72(2):142-9.
[32] Kyaw T, Cui P, Tay C, et al. BAFF receptor mAb treatment ameliorates development and progression of atherosclerosis in hyperlipidemic ApoE(-/-) mice[J]. PLoS One, 2013,8(4):e60430.
[33] Lee M, Park HS, Choi MY, et al. Significance of Soluble CD93 in Type 2 Diabetes as a Biomarker for Diabetic Nephropathy: Integrated Results from Human and Rodent Studies[J]. J Clin Med, 2020,9(5):1394.
[34] Zhao B, Wang D, Liu Y, et al Six-Gene Signature Associated with Immune Cells in the Progression of Atherosclerosis Discovered by Comprehensive Bioinformatics Analyses[J]. Cardiovasc Ther, 2020, 2020:1230513.
[35] Kelly KJ, Liu Y, Zhang J, et al. Renal C3 complement component: feed forward to diabetic kidney disease[J]. Am J Nephrol, 2015,41(1):48-56.
[36] Tanase DM, Gosav EM, Anton MI, et al. Oxidative Stress and NRF2/KEAP1/ARE Pathway in Diabetic Kidney Disease (DKD): New Perspectives[J]. Biomolecules, 2022,12(9):1227.
[37] Jia M, Li Q, Guo J, et al. Deletion of BACH1 Attenuates Atherosclerosis by Reducing Endothelial Inflammation[J]. Circ Res, 2022,130(7):1038-1055.
[38] Fang F, Feng T, Li J, et al. Cathepsin K contributed to disturbed flow-induced atherosclerosis is dependent on integrin-actin cytoskeleton-NF-κB pathway[J]. Genes Dis, 2022,10(2):583-595.
[39] Gudgeon J, Marín-Rubio JL, Trost M. The role of macrophage scavenger receptor 1 (MSR1) in inflammatory disorders and cancer[J]. Front Immunol, 2022,13:1012002.
[40] Uesugi N, Sakata N, Horiuchi S, et al. Glycoxidation-modified macrophages and lipid peroxidation products are associated with the progression of human diabetic nephropathy[J]. Am J Kidney Dis, 2001,38(5):1016-25.
[41] Han SY, Ghee JY, Cha JJ, et al. Upregulation of VSIG4 in Type 2 Diabetic Kidney Disease[J]. Life (Basel), 2022,12(7):1031.
[42] Ding Q, Xing J, Bai F, et al. C1QC, VSIG4, and CFD as Potential Peripheral Blood Biomarkers in Atrial FibrillationRelated Cardioembolic Stroke[J]. Oxid Med Cell Longev, 2023,2023:5199810.
[43] Rotllan N, Zhang X, Canfrán-Duque A, et al. Antagonism of miR148a attenuates atherosclerosis progression in APOBTGApobec- /-Ldlr+/- mice: A brief report[J]. Biomed Pharmacother, 2022,153:113419.
[44] Flyvbjerg A. The role of the complement system in diabetic nephropathy[J]. Nat Rev Nephrol, 2017,13(5):311-318.
[45] Garcia-Arguinzonis M, Diaz-Riera E, Peña E, et al. Alternative C3 Complement System: Lipids and Atherosclerosis[J]. Int J Mol Sci, 2021,22(10):5122.
[46] National Center for Biotechnology Information. PubChem Gene Summary for Gene 6688, SPI1 - Spi-1 proto-oncogene (human) [EB/OL]. Retrieved November 23, 2023 from https://pubchem. ncbi.nlm.nih.gov/gene/SPI1/human.
[47] Wang X, Jiang L, Liu XQ, et al. Identification of Genes Reveals the Mechanism of Cell Ferroptosis in Diabetic Nephropathy[J]. Front Physiol, 2022,13:890566.
[48] Liu C, Zhou Y, Zhou Y, et al. Identification of crucial genes for predicting the risk of atherosclerosis with system lupus erythematosus based on comprehensive bioinformatics analysis and machine learning[J]. Comput Biol Med,2023,152:106388.
[49] National Center for Biotechnology Information. PubChem Gene Summary for Gene 3725, JUN - Jun proto-oncogene, AP-1 transcription factor subunit (human)[EB/OL]. Retrieved November 23, 2023 from https://pubchem.ncbi.nlm.nih.gov/ gene/JUN/human.
[50] Linton MF, Babaev VR, Huang J, et al. Macrophage Apoptosis and Efferocytosis in the Pathogenesis of Atherosclerosis[J]. Circ J, 2016,80(11):2259-2268.
[51] Gao C, Fei X, Wang M, et al. Cardamomin protects from diabetesinduced kidney damage through modulating PI3K/AKT and JAK/STAT signaling pathways in rats[J]. Int Immunopharmacol, 2022,107:108610.
[52] Delgado-Lista J, Perez-Martinez P, Garcia-Rios A, et al. A gene variation (rs12691) in the CCAT/enhancer binding protein α modulates glucose metabolism in metabolic syndrome[J]. Nutr Metab Cardiovasc Dis,2013,23(5):417-23.
[53] Cheng R, Xu X, Yang S, et al. The underlying molecular mechanisms and biomarkers of plaque vulnerability based on bioinformatics analysis[J]. Eur J Med Res, 2022,27(1):212.
[54] Liu J, Duan G, Yang W, et al. Identification of transcription factors related to diabetic tubulointerstitial injury[J]. J Transl Med,2023,21(1):228.
 |
版权:《高科技与产业化》编辑部版权所有 京ICP备12041800号 地址:北京市海淀区中关村北四环西路33号 邮编:100080 联系电话:(010)82626611-6618 传真:(010)82627674 联系邮箱:hitech@mail.las.ac.cn |



